Genómica: conocimiento
Genómica y lucha contra la resistencia a la artemisinina
Uso de la epidemiología genómica para combatir la propagación de parásitos de la malaria resistentes a los fármacos

La malaria es uno de los problemas de salud pública más importantes del mundo. Se estima que hubo 228 millones de casos en 2018, lo que provocó aproximadamente 405,000 muertes1. La malaria es una enfermedad infecciosa transmitida por mosquitos que causa fiebre, cansancio, anemia, vómitos, dolores de cabeza y otros síntomas. En casos graves, puede ocurrir coma, insuficiencia orgánica o la muerte. Es causada por parásitos de la especie Plasmodium. La mayoría de los casos clínicamente graves son causados por la especie Plasmodium falciparum. La incidencia mundial de malaria disminuyó de 2010 a 2018, pero durante los años recientes, la tasa de esa disminución se ha desacelerado sustancialmente, lo que indica que el progreso en la eliminación de la enfermedad se está desacelerando1.
Las terapias combinadas basadas en artemisinina (Artemisinin-based combination therapies, ACT) han contribuido a las recientes reducciones de la morbilidad y la mortalidad. Los derivados de la artemisinina se descubrieron en los años 70 del siglo XX y son potentes antipalúdicos. Debido a que tienen semividas cortas, se usan junto con fármacos de acción más prolongada, como amodiaquina, piperaquina, mefloquina, sulfadoxina/pirimetamina o lumefantrina. Estas terapias combinadas eficaces se han convertido en la piedra angular del tratamiento de la malaria en áreas endémicas. El uso de ACT ha sido responsable de reducciones sustanciales en la carga de parásitos y muertes en África2.
Los diferentes fármacos antipalúdicos actúan a través de una variedad de mecanismos, que incluyen el bloqueo de las vías de desintoxicación de los parásitos, la interferencia con el metabolismo del folato y la inhibición de la función mitocondrial. Los derivados de la artemisinina tienen un mecanismo de acción complejo que puede matar al parásito en una variedad de etapas del ciclo de vida al causar un daño generalizado en las proteínas del parásito. La artemisinina y sus derivados se activan mediante la exposición al hemo libre, un compuesto que contiene hierro y que compone la parte no proteica de la hemoglobina. La exposición al hemo da como resultado la división en la forma activa de la artemisinina, dihidroartemisinina (DHA). Luego, la DHA causa un daño irreversible a las proteínas del parásito y la acumulación de estas proteínas desdobladas y dañadas. La acumulación de proteínas dañadas activa la respuesta al estrés del retículo endoplásmico y conduce a la muerte del parásito3.
La creciente prevalencia de P.
falciparum resistente a la artemisinina en gran parte del sudeste asiático amenaza con desestabilizar el control de la malaria en todo el mundo
La resistencia a los fármacos de Plasmodium, la capacidad de los parásitos para sobrevivir y reproducirse, a pesar de la administración de antipalúdicos, es un problema recurrente en los esfuerzos de control y erradicación. Ha surgido resistencia a todos los antipalúdicos conocidos, y los parásitos resistentes emergentes son una amenaza significativa para el progreso contra la malaria. La reciente aparición de resistencia a las ACT en el sudeste asiático es motivo de especial preocupación. Algunos parásitos ahora pueden tolerar la artemisinina en sus etapas iniciales de vida al entrar en un período de latencia y luego reanudar el crecimiento después de que el tratamiento con el fármaco ha disminuido, lo que genera tasas más bajas de eliminación del parásito4. La evidencia de resistencia a la artemisinina se observó por primera vez en 2008 en Camboya, y ahora los parásitos resistentes se están extendiendo por toda la región del Gran Mekong5. Por ejemplo, el tratamiento con dihidroartemisinina-piperaquina tuvo tasas de fracaso entre el 14.3 % y el 46.3 % en estudios realizados entre 2015 y 2017 en Vietnam y la artesunato-mefloquina reemplazó a la dihidroartemisinina-piperaquina como el tratamiento de primera línea recomendado en Camboya, después de que se observaron altas tasas de fracaso6. La propagación de la resistencia a África, desde el sudeste asiático a través de Bangladesh y la India, es motivo de preocupación porque esta ruta fue la fuente de la resistencia ahora generalizada a otro antipalúdico, la cloroquina, en África7. La resistencia a las ACT en África podría ocasionar epidemias importantes y poner en peligro los avances recientes8. Prevenir el establecimiento de resistencia a las ACT en África representa un desafío para los esfuerzos actuales de control de la malaria.
La disminución de la eficacia de la artemisinina en los parásitos jóvenes se asocia con mutaciones en Kelch13, una proteína ubicada en el cromosoma 13. Se desconoce el mecanismo exacto de esta resistencia, pero las mutaciones en Kelch13 podrían ayudar a los parásitos a resistir el daño de la artemisinina al proteger a las proteínas de dicho daño, prevenir la acumulación de proteínas dañadas o disminuir la cantidad de hemo libre y
reducir la activación de la artemisinina en sí. Además, las mutaciones de Kelch13 podrían retrasar el crecimiento del parásito y permitir que este imite una fase inactiva y resista el daño4.Kelch13 fue identificado como un marcador molecular en la resistencia a la artemisinina en 20149. La identificación inicial de marcadores de resistencia molecular, como mutaciones en Kelch13, implica el uso de estudios de asociación de genoma completo (genome-wide association study, GWAS) y otros estudios de asociación genética para (1) encontrar posiciones en el cromosoma que están bajo una fuerte presión selectiva; e (2) identificar mutaciones específicas asociadas con la resistencia2, 10. Una vez que se identifica un marcador, debe validarse mediante una variedad de estudios de laboratorio. Se han identificado más de 20 polimorfismos de un solo nucleótido (SNP) diferentes en Kelch13 que confieren resistencia a la artemisinina, y estos polimorfismos son el resultado de eventos mutacionales independientes10,11.
Los datos genómicos, como los datos de secuenciación de genoma completo (whole genome sequencing, WGS), se pueden utilizar para rastrear la prevalencia y la propagación de estas cepas resistentes proporcionando información detallada sobre la estructura de la población del parásito. Un estudio del Proyecto Comunitario sobre P. falciparum de MalariaGEN (Red de Epidemiología Genómica de la Malaria) analizó los datos de secuenciación de más de 3,000 muestras clínicas de 23 países de África y el sudeste asiático. El estudio determinó que las mutaciones de kelch13 aparecen con poca frecuencia en África y se han producido independientemente de los parásitos del sudeste asiático. Estas mutaciones se mantienen en niveles bajos en la población y no han sufrido el mismo grado de presión selectiva que las del sudeste asiático, lo que demuestra que factores más allá de la mutación por sí solos afectan la propagación de la resistencia. Los patrones de tratamientos farmacológicos y otros factores ambientales también determinan cómo se propagan las mutaciones, y los datos genómicos son una herramienta esencial para tratar de desentrañar estos complejos sistemas. Estos hallazgos demuestran la importancia de aplicar datos genómicos para comprender por qué se propaga la resistencia y garantizar que la malaria resistente a la ACT no se convierta en un problema importante en África12.
Otro estudio reciente analizó más de 2,000 genomas del Proyecto Comunitario sobre P. falciparum de MalariaGEN recopilados entre 2007 y 2018 para reunir datos epidemiológicos a escala fina sobre la cepa del parásito KEL1/PLA1 que es resistente al tratamiento con dihidroartemisinina-piperaquina. Los parásitos KEL1/PLA1 se han extendido por el sudeste asiático oriental desde 2015, y han reemplazado a muchas de las poblaciones de parásitos locales anteriores. Varios grupos de estos parásitos se han propagado en oleadas independientes. Es probable que la exposición a la dihidroartemisinina-piperaquina después de que surgiera la resistencia ocasionara un aumento de la presión del fármaco y resultara en poblaciones de parásitos con mutaciones adicionales que son más resistentes al tratamiento13. Este estudio destaca la importancia de los datos genéticos para informar los esfuerzos de vigilancia y prevenir la aparición de especies resistentes a varios fármacos.
Hasta la fecha, la mayoría de las técnicas de vigilancia genómica y molecular se han empleado principalmente en contextos de investigación y no se han integrado por completo en las prácticas de vigilancia de rutina14. Los avances recientes en las tecnologías de secuenciación y las reducciones en los costos asociados están haciendo que las técnicas genómicas sean más accesibles. El costo de la secuenciación del ADN por megabase sin procesar, 1 millón de nucleótidos, ha disminuido de $10,000,000 en 2001 a $0.01 en 2019, y las disminuciones más pronunciadas en el costo ocurrieron después de 2008, cuando muchos centros pasaron a la secuenciación de próxima generación desde la secuenciación tradicional de Sanger15. La capacidad de obtener la WGS a partir de una gota de sangre seca (dried blood spot, DBS), donde las muestras de sangre se transfieren y secan en papel de filtro, fue un avance significativo en la traducción de la tecnología genómica a los esfuerzos de vigilancia de la malaria en el mundo real. Las DBS se pueden adquirir con recursos y personal mínimos y se pueden almacenar sin refrigeración, lo que las convierte en una forma ideal de recolección de muestras en entornos con recursos limitados16. Si bien las técnicas de secuenciación aún requieren inversiones en infraestructura, potencia informática y personal capacitado, el desarrollo de laboratorios de referencia nacionales y subregionales centralizados podría ser de ayuda.
La integración de datos epidemiológicos, moleculares y genómicos podría mejorar sustancialmente la vigilancia de la malaria en entornos de eliminación y
reducir estos costos. Además, la WGS no es necesaria en todos los contextos. Una vez que se han identificado nuevas variantes genéticas y se han validado los marcadores moleculares, se pueden usar métodos de menor costo que se dirigen a regiones específicas del genoma. El intercambio de datos y muestras también es esencial. Las iniciativas como MalariaGEN han proporcionado ejemplos de esfuerzos de colaboración exitosos14. Todavía hay tiempo para prevenir la propagación de la resistencia a la ACT a escala mundial, pero estos esfuerzos requerirán la integración eficiente de datos epidemiológicos, moleculares y genómicos en la vigilancia de rutina.
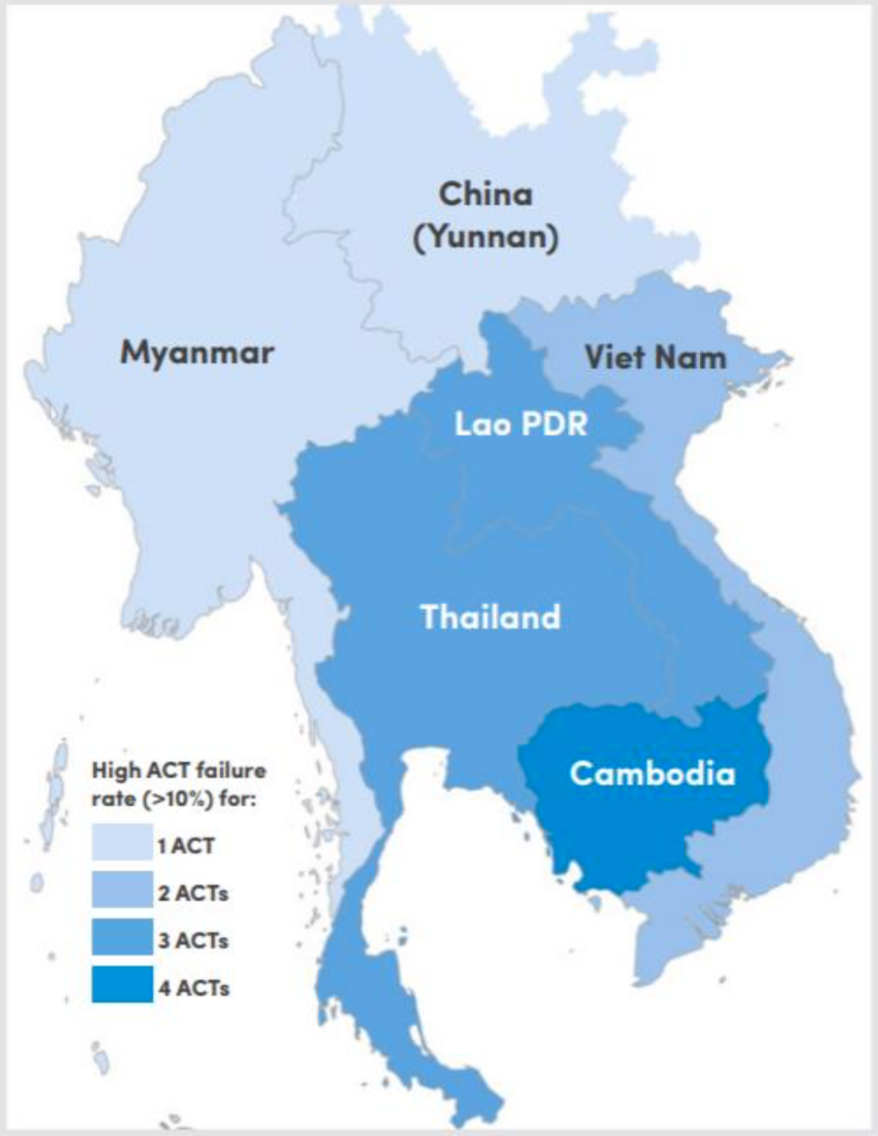
Figura 1. Clasificación de los países del sudeste asiático por número de ACT que fallaron (>10 % de fracaso en el tratamiento) después de 2010. (Fuente: Informe de estado de la OMS sobre la resistencia a la artemisinina y la eficacia de la ACT, diciembre de 2018).
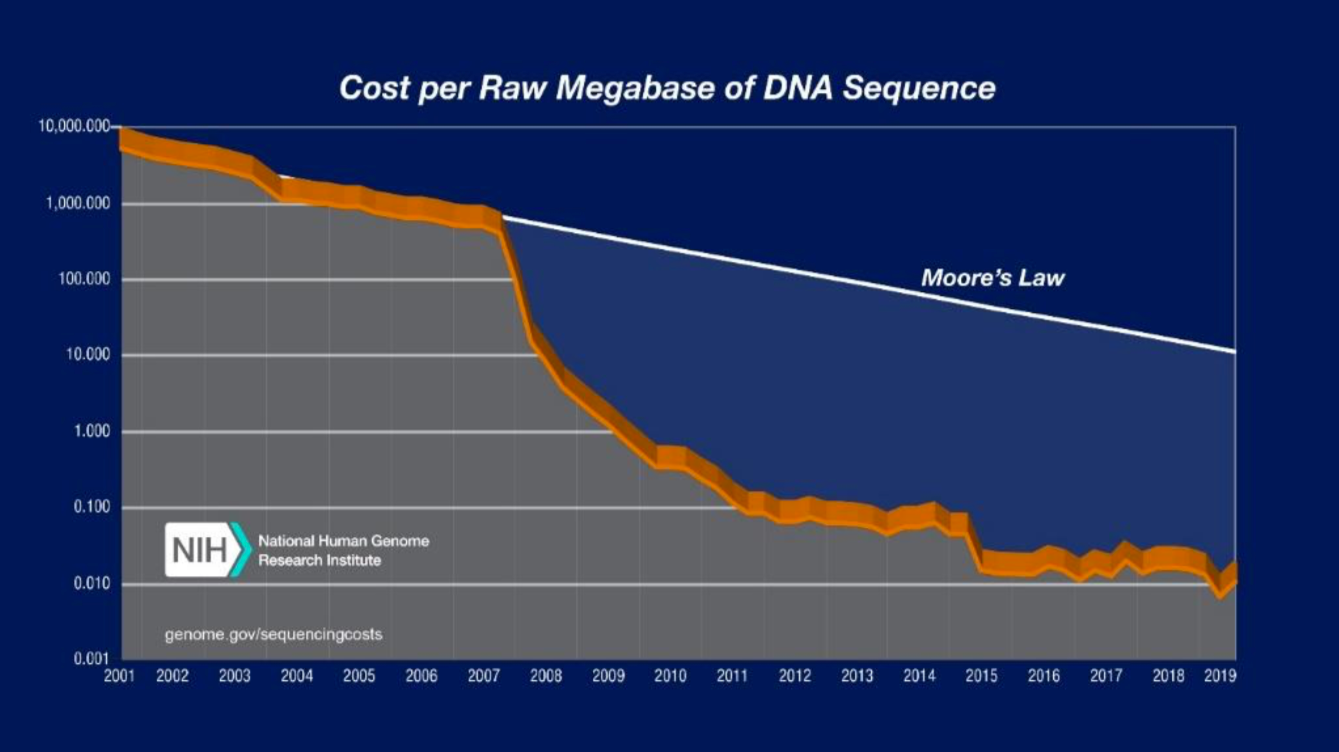
Figura 2. Disminución de los costos de secuenciación de 2001 a 2019. El gráfico muestra los costos asociados con la secuenciación de ADN de 2001 a 2019. La ley de Moore describe una tendencia en la industria del hardware informático en la que la potencia informática se duplica cada dos años. Por lo general, se considera que la tecnología que avanza al ritmo de la ley de Moore avanza al mismo ritmo. Los costos de secuenciación han disminuido rápidamente, superando la ley de Moore de forma significativa. (Fuente: costos de la secuenciación del ADN: datos).
Acerca del autor

Emma Rowley es becaria de posgrado en el Laboratorio de Investigación de la Malaria y Vectores del Instituto Nacional de Alergias y Enfermedades Infecciosas, donde está investigando Plasmodium vivax, un parásito que causa la malaria. Se graduó de la Universidad Wake Forest en 2019 con una licenciatura en Biología.