Genómica: conocimiento

¿La modificación de los genes de la respuesta inmunitaria puede evitar que los pacientes con alo-TCMH contraigan el SDRA?
El síndrome de dificultad respiratoria aguda (SDRA) es potencialmente mortal al permitir que el líquido de los pulmones se acumule en los alvéolos, pequeños sacos de aire en los pulmones que intercambian oxígeno y dióxido de carbono. Esto evita que entre en el torrente sanguíneo una cantidad suficiente de oxígeno necesaria para que los órganos funcionen correctamente[2]. El SDRA tiene importantes consecuencias a largo plazo, como deterioro físico y cognitivo y una alta tasa de mortalidad entre el 30 y el 40 % como causa de muerte en las unidades de cuidados intensivos para adultos[3].
Introducción
Un artículo de investigación respiratoria publicado por BMC compara los patrones de expresión de ARN en sangre oxigenada entre pacientes con SDRA que han recibido un trasplante alogénico de células madre hematopoyéticas (alo-TCMH), un tratamiento para el cáncer de sangre, y aquellos que no lo han recibido[1]. La identificación de patrones de expresión de ARN nos ayuda a comprender qué genes aumentan o reducen y si ciertos genes están modulados. El alo-TCMH es un procedimiento en el que los pacientes con cáncer con un sistema inmunitario deficiente son tratados con células especializadas[4]. Estas células especializadas crecen hasta convertirse en células sanguíneas y desarrollan un nuevo sistema inmunitario después de que la quimioterapia destruye el sistema inmunitario anterior[5]. Los pacientes con cáncer de sangre sometidos a alo-TCMH son un subgrupo importante con mayor riesgo de sufrir el SDRA y altas tasas de mortalidad. El objetivo de este estudio es identificar las diferencias en la expresión génica entre sujetos con SDRA que recibieron y no recibieron alo-TCMH. Al identificar estas diferencias podemos apuntar a regiones genéticas específicas para prevenir el desarrollo de SDRA en pacientes con alo-TCMH.
(...) subrayar la necesidad de una mejor comprensión de estas diferentes poblaciones.
Método
Los investigadores utilizaron muestras de sangre de 11 pacientes con SDRA para perfilar la expresión génica que se tomaron durante los 5 días posteriores al ingreso en la Unidad de Cuidados Intensivo (UCI), y los sujetos fueron seleccionados por edades, sexo y raza similares para reducir la heterogeneidad de la muestra y producir resultados confiables. Estos 11 pacientes consistían en 5 personas que se habían sometido a un alo-TCMH y 6 personas que no habían recibido el trasplante. El estudio incluyó a 12 pacientes adicionales con sepsis por TCMH y sepsis sin TCMH, con el fin de identificar diferencias entre los grupos de SDRA que no fueron causadas por el estado del TCMH. Se comparó la expresión génica de pacientes con SDRA y pacientes con SDRA posterior al alo-TCMH mediante secuenciación de ARN. La secuenciación de ARN se realiza al aislar y purificar el ARN, lo que convierte el ARN en ADNc, también conocido como ADN complementario, que es una copia procesada del ARN, y al analizar las secuencias resultantes del ADNc[6]. Esto muestra el transcriptoma de los patrones de expresión génica codificados dentro del ARN[7].
El diseño general del estudio se muestra a continuación:
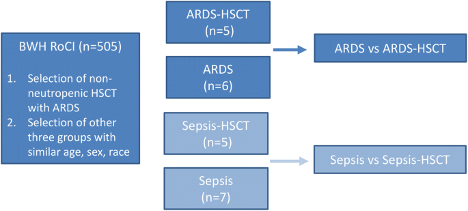
También se incluyeron sujetos con sepsis para identificar diferencias entre los grupos con SDRA que no fueron causadas por el estado del TCMH.
Resultados
A través de la secuenciación de ARN se identificaron 687 genes expresados diferencialmente entre SDRA-TCMH y SDRA. 520 de estos genes aumentaron y 167 se redujeron. Después de filtrar los genes también expresados en sepsis-TCMH y sepsis, se descubrió que 483 genes eran exclusivos de SDRA-TCMH. Curiosamente, algunos de los genes expresados diferencialmente con las desigualdades más fuertes en las vías, como IFI44L, OAS3, LY6E, OAS2 y USP18, estaban relacionados con respuestas inmunes a patógenos y señalización del interferón tipo I, todos los cuales aumentaron en el SDRA. Aunque los interferones de tipo I aumentan la resistencia celular a la infección viral al iniciar respuestas inflamatorias pulmonares, esta inflamación, a veces, contribuye con la gravedad de la enfermedad[8].
La modulación de las vías de señalización del interferón representa un enfoque de tratamiento para los sujetos con TCMH (...).
Lo que sugieren estos resultados es que existe una desregulación inmunitaria (funcionamiento deficiente del sistema inmunitario) que hace que los pacientes sean más susceptibles al SDRA-TCMH. Se encontró una expresión sanguínea alta y baja de MX1, IFIT1 e ISG15, un conjunto de genes estimulados por interferón, en pacientes con resultados nefastos. En otro estudio, el análisis de la expresión de genes de monocitos, un tipo de glóbulo blanco en sangre periférica, reveló un aumento de la señalización del interferón en pacientes que tenían mejores resultados, en comparación con pacientes que tenían resultados desfavorables[9]. Esto sugiere que la modulación de las vías de señalización del interferón podría potencialmente convertirse en un método de tratamiento más dirigido a sujetos con TCMH. Este estudio y la literatura existente sugieren que al modular las vías de señalización del interferón, la respuesta inmunitaria que desencadena el desarrollo del SDRA después de un alo-TCMH podría modificarse, lo que evita que pacientes con alo-TCMH desarrollen el SDRA.
Conclusión
Estudios anteriores identificaron firmas de expresión génica en pacientes con sepsis que estaban desarrollando el SDRA y descubrieron que los pacientes con SDRA tenían aumentos drásticos en las vías de respuesta inmunitaria, un conjunto de procesos que desencadena una respuesta inmunitaria ante una sustancia extraña[10], en comparación con personas sanas. Sin embargo, ninguno de estos estudios caracterizó subgrupos de SDRA, lo que hace que este estudio sea importante porque la caracterización de subgrupos puede revelar cambios en la expresión génica que dan como resultado síntomas clínicos similares.
Este estudio identificó diferencias en la expresión génica del SDRA y SDRA-TCMH, lo que proporciona un posible método de tratamiento del SDRA y demuestra la necesidad de identificar subgrupos del SDRA para su tratamiento. Los hallazgos revelaron cambios en la respuesta inmunitaria y las vías de señalización del interferón, lo que sugiere que la señalización del interferón desregulada es la causa de la lesión pulmonar después del alo-TCMH. Tanto los estudios anteriores como este estudio apoyan la posibilidad de modificar la inmunodeficiencia para que los pacientes que se someten a alo-TCMH no corran riesgo de lesión pulmonar y SDRA. La investigación adicional del tratamiento mediante la modificación de la inmunodeficiencia es una gran esperanza para el futuro del SDRA.
Referencias
- Englert, Joshua. “Whole Blood RNA Sequencing Reveals a Unique Transcriptomic Profile in Patients with ARDS Following Hematopoietic Stem Cell Transplantation.” Respiratory Research, BioMed Central, 1 Jan. 1970.
- “ARDS.” Mayo Clinic, Mayo Foundation for Medical Education and Research, 13 June 2020.
- Bellani, Giacomo. “Epidemiology, Patterns of Care, and Mortality for Patients With Acute Respiratory Distress Syndrome in Intensive Care Units in 50 Countries.” JAMA, U.S. National Library of Medicine, 2016.
- “What Is Hematopoietic Stem Cell Transplantation?” Medical News Today, MediLexicon International, 2017.
- Schley, David. “7 Things You Need to Know about HSCT.” Multiple Sclerosis Society UK, 2018.
- Cresko Lab. “Seqlopedia.”.
- MacKenzie, Ruairi. “RNA-Seq: Basics, Applications and Protocol.” Genomics Research from Technology Networks, 2018.
- Makris, Spyridon. “Type I Interferons as Regulators of Lung Inflammation.” Frontiers in Immunology, Frontiers Media S.A., 10 Mar. 2017, www.ncbi.nlm.nih.gov/pmc/articles/PMC5344902/.
- Morrell, Eric. “Peripheral and Alveolar Cell Transcriptional Programs Are Distinct in Acute Respiratory Distress Syndrome.” American Journal of Respiratory and Critical Care Medicine, 2017, www.atsjournals.org/doi/10.1164/rccm.201703-0614LE.
- The Jackson Laboratory. “Immune Response-Regulating Signaling Pathway.” Immune Response-Regulating Signaling Pathway Gene Ontology Term (GO:0002764), 2020.
Acerca del autor
Beatrice Park es una estudiante de 15 años que asiste a la escuela secundaria Northside College Preparatory. Aspira a realizar investigaciones en el campo de respiración y está fascinada por la función de la genómica en las enfermedades y síndromes respiratorios.