Genómica: conocimiento

¿Las bacterias genéticamente modificadas pueden resolver la crisis de contaminación plástica?
Estudio de desechos plásticos con enzimas bacterianas que comen plástico
El plástico es una innovación que ha impregnado todos los aspectos de la vida moderna, pero la dependencia humana hacia el plástico tiene un precio alto. El plástico permanece en el medio ambiente durante milenios, se descompone en microplásticos que llegan al cuerpo humano y amenazan gravemente la salud de la humanidad y nuestro entorno. Sin embargo, la genómica y la biotecnología son la clave para resolver este problema urgente. Como sugiere su nombre, las bacterias que comen plástico son organismos recientemente descubiertos que son capaces de digerir plásticos creados por el hombre. Junto con el análisis genómico y la ingeniería genética, estos microbios tienen el potencial de producir una solución revolucionaria a la crisis de contaminación plástica.
¿Qué son las bacterias que comen plástico?
Las bacterias que comen plástico son una clasificación de microorganismos que pueden digerir polímeros sintéticos. El plástico no se puede descomponer de manera eficiente y permanece en el medio ambiente durante milenios; solo se produjo en masa el siglo pasado y los descomponedores no se han adaptado lo suficientemente rápido para digerir su compleja estructura molecular. Sin embargo, el descubrimiento de bacterias que comen plástico niega la creencia anterior de que los descomponedores no pueden digerir el plástico de forma natural. Las habilidades de las bacterias que comen plástico y otros organismos son muy singulares en un contexto genómico y evolutivo, ya que demuestran la aparición de un nuevo comportamiento que no existía antes. Sin embargo, su capacidad para consumir polímeros artificiales se debe a las diversas enzimas que producen en sus sistemas digestivos.
"Las bacterias que comen plástico son una clasificación de microorganismos que pueden digerir polímeros sintéticos".
¿Qué tipos de bacterias y enzimas que comen plástico existen?
Las bacterias que comen plástico se pueden clasificar en dos grupos: bacterias que se alimentan de nailon y polietilentereftalato (polyethylene terephthalate, PET) (dos tipos diferentes de plástico). El primer tipo se descubrió en 1975 en los estanques de drenaje de aguas residuales de una planta japonesa de nailon. La llamaron Flavobacterium sp. K172 y no existía antes de la producción masiva de nailon, y se descubrió que sobrevivían únicamente en dímeros cíclicos como el ácido 6-aminohexanoico, un subproducto de la producción de nailon-6. También descubrieron que la hidrolasa del dímero cíclico del ácido 6-aminohexanoico y la hidrolasa del oligómero lineal del ácido 6-aminohexanoico, las enzimas responsables de la digestión del nailon, no podían digerir ningún otro compuesto, excepto subproductos artificiales del nailon3. En conjunto, estas enzimas digestivas bacterianas se conocieron como nailonasas. Estudios posteriores sugieren que las nailonasas del gen, como la hidrolasa del ácido 6-aminohexanoico, surgieron de la combinación de una duplicación de genes y una mutación de cambio de marco aleatorio y que estos cambios en múltiples genes contribuyeron con la evolución de la cepa. 4
La otra forma de bacterias que comen plástico son las bacterias que se alimentan de PET. La primera cepa de bacterias que comen PET se aisló en 2016 de una planta en la ciudad de Sakai, Japón. La bacteria gramnegativa con forma de bastón, denominada Ideonella sakaiensis, está estrechamente relacionada con las especies de Ideonella dechloratans e Ideonella azotifigens que no comen plástico mediante la secuenciación de genes de ARN y el análisis filogenético5. Las bacterias que comen PET utilizan dos enzimas para digerir PET: PETasa y MHETasa. La bacteria secreta ambas enzimas simultáneamente que descomponen el PET en sustancias ambientalmente benignas. Una colonia de I. sakaiensis puede degradar por completo una botella de agua de plástico de baja calidad en seis semanas. Los productos de PET de mayor calidad requerirían calentamiento y enfriamiento para debilitarlos antes de que las bacterias puedan comenzarlos a comer7.
También se han descubierto enzimas independientes capaces de digerir PET. En 2012, los investigadores encontraron cutinasa de abono de ramas y hojas (leaf-branch compost cutinase, LLC). Esta enzima evolucionó para romper la capa protectora cerosa de las hojas de muchas plantas, pero se adaptó a los enlaces químicos del PET. Sin embargo, la enzima solo puede funcionar después de días a una temperatura específica, cuando lentamente comienza a romper los enlaces del polímero10.
¿Cómo actúan las nailonasas y las PETasas?
El mecanismo detrás de todas las bacterias y enzimas que digieren plástico es la rotura de los enlaces químicos, o ésteres, en un polímero sintético como el nailon o el PET. Esto descompone la molécula compleja en monómeros más simples que este organismo puede digerir. La nailonasa NylB, codificada por el gen nylB, solo puede degradar dímeros de nailon y oligómeros cortos. Esto significa que la nailonasa solo puede digerir subproductos de desecho del nailon.
El PET tiene una estructura cristalina, por lo que es recalcitrante a la despolimerización catalítica, ya que los enlaces éster son demasiado fuertes. La PETasa funciona convirtiendo el PET en ácido mono (2-hidroxietil) tereftálico (MHET), con trazas de ácido tereftálico (terephthalic acid, TPA) y bis (2-hidroxietil)-TPA como productos secundarios. La MHETasa trabaja de la mano con la PETasa, convirtiendo aún más el MHET en sus dos monómeros, TPA y etilenglicol (ethylene glycol, EG), que luego son digeribles por I. sakaiensis y sirven como fuente de energía.
Al igual que la PETasa en bacterias, la LLC funciona cortando los enlaces entre los dos componentes básicos del PET: tereftalato y etilenglicol. Pero la LLC solo puede introducirse en los enlaces del polímero y descomponerlos después de unos días de trabajo a 65 °C, la temperatura a la que el PET se ablanda, después de lo cual se desnaturaliza naturalmente (figura 1).
¿Cómo podemos usar las enzimas que comen plástico y la edición de genes para combatir la contaminación plástica?
Los científicos pudieron inducir otras especies de bacterias, como Pseudomonas aeruginosa y E. coli, para desarrollar la capacidad de descomponer los mismos subproductos de nailon de la nailonasa en un laboratorio al obligarlas a vivir en un entorno sin otra fuente de nutrientes y transferencia de plásmidos4. Estos ejemplos muestran que la nailonasa se puede aprovechar para utilizarla en una variedad de especies de bacterias económicas y fácilmente accesibles que luego se pueden utilizar en masa para combatir la contaminación plástica.
La P. aeruginosa también puede digerir el PET y otros plásticos como polietileno, polipropileno y muchos más con diversos grados de eficiencia. La PETasa también tiene atributos que la convierten en un método plausible para combatir el plástico: está diseñada físicamente para adherirse a superficies del PET y funciona a 30 °C, lo que la hace adecuada para el reciclaje en biorreactores.
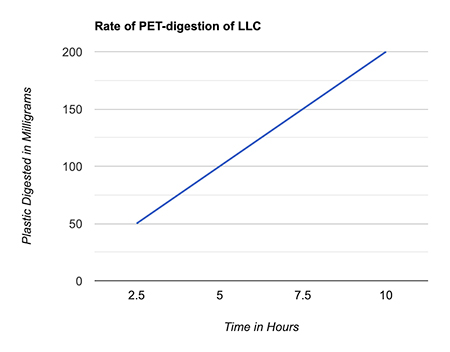 Figura 1. Velocidad a la que la enzima LLC consume el PET en una magnitud de miligramos por hora (creada por la autora JiaJia Fu usando Google Sheets).
Figura 1. Velocidad a la que la enzima LLC consume el PET en una magnitud de miligramos por hora (creada por la autora JiaJia Fu usando Google Sheets).¿Por qué las enzimas que comen plástico son mejores que las soluciones existentes para gestionar los residuos plásticos?
Solo el 9 % de todos los plásticos producidos se reciclan debido al alto costo de descomponer químicamente el plástico en relación con sus costos de producción económicos y eficientes1. Dado que el reciclaje es costoso y consume recursos, la mayoría del plástico va a los vertederos o al medio ambiente. La mayoría de las soluciones propuestas para gestionar los desechos plásticos solo se enfocan en las secuelas, cuando el plástico ingresa al medio ambiente y se convierte en contaminación. Aparte de intentar reducir la producción de plástico y su entrada al medio ambiente (medidas físicas como redes para recolectar plástico en el océano), los humanos no han encontrado una forma de eliminar realmente los desechos en sí hasta ahora. La edición de genes mediante repeticiones palindrómicas cortas agrupadas y regularmente interespaciadas (clustered regularly interspaced short palindromic repeats, CRISPR) y otras técnicas genómicas también es la clave para producir una solución viable y asequible mediante la utilización de mecanismos naturales. Incluso si una cepa o enzima bacteriana específica solo puede digerir un tipo de plástico o descompone su componente respectivo con demasiada lentitud, situaciones que son inaplicables en un contexto comercial, los investigadores pueden alterarlas para hacer que el proceso sea más rápido, más eficiente y efectivo. Mediante el análisis de la estructura cristalina de la enzima y la identificación de los aminoácidos clave, los investigadores aislaron una enzima mutante 10,000 veces más eficiente para romper el PET que la LLC natural. El equipo descubrió que podía descomponer el 90 % de 200 gramos de PET en 10 horas. Convirtieron el 90 % de ese mismo plástico en sus materiales de partida y resintetizaron el PET a partir de los compuestos de tereftalato y etilenglicol; el PET reciclado era tan fuerte como el plástico convencional. El éxito de sus pruebas ha llevado a ampliar la tecnología y planea abrir una planta de demostración el próximo año.
"La mayoría de las soluciones propuestas para gestionar los desechos plásticos solo se enfocan en las secuelas, cuando el plástico ingresa al medio ambiente y se convierte en contaminación".
¿Cuáles son los posibles impactos y consecuencias de las enzimas que comen plástico?
Gracias a la genómica y la biotecnología, el potencial de un mecanismo biológico único puede realizarse plenamente e incluso comenzar a aplicarse a gran escala. Los impactos y la aplicación de una tecnología o proceso desarrollado a partir de bacterias y enzimas que comen plástico son inconmensurables. Las enzimas que comen plástico podrían generar un proceso confiable de manejo de desechos plásticos industriales, un reciclaje verdaderamente efectivo, plástico oceánico y microplásticos que eliminan sueros, o incluso en el hogar, contenedores de abono que consumen desechos plásticos.
Los posibles impactos de la utilización de bacterias que comen plástico son innegables, pero también lo son los posibles contratiempos y consecuencias de implementar esta tecnología.
Aunque la manipulación genética puede desbloquear el potencial de la nailonasa, en la actualidad no se puede utilizar a gran escala, ya que no puede digerir el nailon comercial, sino los productos de desecho que ya se han degradado en oligómeros cortos. La viabilidad económica de la LLC tampoco está clara. Solo puede descomponer el PET puro y no los tintes y otros plásticos mezclados en productos plásticos. La introducción de vida editada genéticamente en el entorno general podría tener consecuencias imprevistas. No hay forma de predecir cómo reaccionaría una bacteria que se alimenta de plástico por sí misma, ni siquiera considerando la manipulación genética, con un nuevo ecosistema como el océano o un vertedero. Las formas en que las bacterias podrían mutar e interactuar con su entorno son demasiado impredecibles para liberarlas sin la investigación y las consideraciones adecuadas.
Aunque existen preocupaciones y riesgos relacionados con el desarrollo de una tecnología de este tipo, es indudable que las enzimas bacterianas que comen plástico pueden liderar los esfuerzos para apaciguar el desastre ambiental al que se enfrenta el mundo. Mediante el uso de la genómica, la biología de vanguardia y la naturaleza, estamos un paso más cerca de resolver la crisis de la contaminación plástica y forjar una sociedad, un planeta y un futuro más saludables.
"Mediante el uso de la genómica, la biología de vanguardia y la naturaleza, estamos un paso más cerca de resolver la crisis de la contaminación plástica y forjar una sociedad, un planeta y un futuro más saludables".
Acerca del autor
JiaJia Fu es una estudiante de primer año en Whittle School and Studios. Nació y creció en Nueva Jersey, le apasiona la ciencia y la naturaleza y aspira crear soluciones innovadoras para problemas ecológicos como la contaminación plástica, la agricultura intensiva y el cambio climático a través de la biotecnología.