La variación reguladora es la variación de la secuencia de ADN que se produce en regiones genómicas no codificantes que influyen en la expresión génica. Aproximadamente el 93 % de las variantes del estudio de asociación de genoma completo (genome-wide association study, GWAS) relacionado con enfermedades y rasgos se encuentran en regiones no codificantes del genoma, y aproximadamente el 20 % se ubica a entre 100 kb y 1 Mb de cualquier secuencia codificante. Por lo tanto, es importante investigar cómo variantes no codificantes influyen en enfermedades y en rasgos y los vinculan a regiones reguladoras, lo que determina cómo afectan la expresión génica sobre largas distancias y cuáles son sus genes objetivo.
“(...) el genoma está fuertemente empaquetado en el núcleo y adquiere una estructura tridimensional en el espacio (...)”.
Si bien tendemos a pensar en el genoma como una secuencia lineal de As, Ts, Cs y Gs, en realidad está empaquetado en el núcleo y adquiere una estructura tridimensional en el espacio. Elementos reguladores no codificantes, como los potenciadores, pueden regular el nivel de expresión de un gen a cientos de kilobases de distancia mediante un bucle cromosómico que puede traer elementos reguladores distales en proximidad tridimensional a los genes objetivo (figura 1). Puede ocurrir una variación en el elemento regulador que puede afectar su capacidad para regular la expresión génica, a menudo, al afectar la unión de un factor de transcripción (figura 1). Por ejemplo, un estudio estableció evidencia de que una región reguladora que contiene la variante rs378854 asociada al cáncer de próstata forma un bucle e interactúa con el oncogén PVT1 a una distancia de 500,000 pares de bases. En el alelo de riesgo de esta variante se reduce la unión de un factor de transcripción represivo conocido como YY1 y, por lo tanto, aumenta la expresión de PVT1. En una escala más amplia, las variantes no codificantes tienden a colocalizarse en elementos reguladores. En un estudio sobre variantes no codificantes en la enfermedad inflamatoria intestinal (inflammatory bowel disease, IBD), 92 de 163 de las variantes no codificantes se encuentran en elementos reguladores3. Al analizar estos elementos reguladores en el contexto del genoma 3D, el mismo grupo de investigadores pudo conectar estos elementos con genes posteriores. Se sabe que algunos de estos genes están implicados en la IBD y algunos podrían ser genes candidatos nuevos.
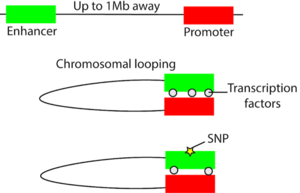
Figura 1: Interrupción del bucle cromosómico
Sin embargo, interrumpir las interacciones entre las regiones reguladoras y los promotores no es la única forma en que la variación no codificante puede influir en la expresión génica. La variación no codificante también puede alterar unidades más grandes de la organización de cromatina, conocidas como dominios asociados topológicamente (topologically associated domains, TAD). Un TAD es un tramo de secuencia de ADN (hasta 1 Mb) donde es más probable que las regiones internas interactúen entre sí que con las regiones externas. En otras palabras, existe un alto grado de bucle cromosómico dentro de los límites del TAD. Si una variante interrumpe el límite del TAD, entonces los potenciadores que normalmente solo interactúan con sus genes objetivo de tipo salvaje dentro del TAD estarían abiertos para influir en la expresión de genes fuera del TAD (figura 2). A veces, esto se denomina "secuestro de potenciadores". Un ejemplo clásico de secuestro de potenciadores es la polidactilia, una condición que resulta en más de cinco dedos en las manos o pies. La polidactilia es causada por una eliminación de un límite del TAD, lo que da como resultado un grupo de potenciadores asociados con el desarrollo de las extremidades que interactúan con genes con los que no interactuarían si el límite hubiera estado en su lugar5. Dependiendo de qué límites del TAD se eliminen, los potenciadores pueden interactuar con diferentes genes que conducen a diferentes fenotipos, como el síndrome F o la braquidactilia.
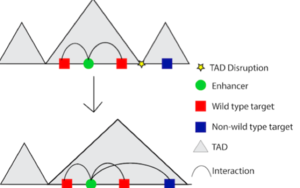
Figura 2: Interrupción de los límites del TAD
Están surgiendo nuevas tecnologías y enfoques informáticos para detectar interacciones cromosómicas en todo el genoma, lo que genera oportunidades nuevas y emocionantes para explorar la función de la variación reguladora en el contexto del genoma 3D. En el futuro, nuestra atención debe enfocarse en comprender la función de la enfermedad no codificante y las variantes asociadas a rasgos que se han identificado en estudios anteriores del GWAS. Deberíamos utilizar estas tecnologías y enfoques informáticos para mapear variantes del GWAS no codificantes en sus genes objetivo. También es importante investigar los efectos de la variación no codificante en los genes objetivo en el contexto de la enfermedad o rasgo. Comprender la relación entre el genoma 3D y la variación reguladora nos permitirá interpretar muchas de las variaciones del GWAS conocidas al vincular las variantes directamente a genes candidatos nuevos y conocidos para una amplia gama de enfermedades y rasgos.